以下の記事でOpenCVを用いて細胞の輪郭検出を行いましたが、今回は特殊なゲルに包まれた細胞を検出していきます。
目次
解析対象(生データ)
今回の解析対象は、特殊なゲル(SeaPlaque)中にトラップされた細胞の画像です。
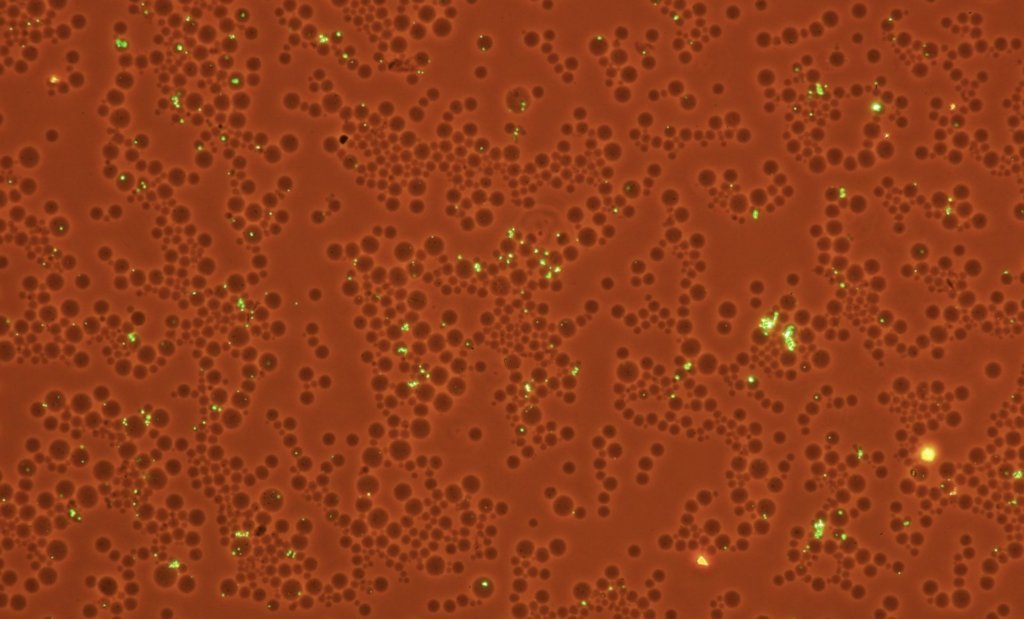
緑色の光がSYBR Green Iによる蛍光です。
また、泡のようなものがゲルで実際は球体になっていますが、顕微鏡写真では微分干渉で見ていないので平面的に見えます。
細胞の検出
初めに、細胞を検出するために細胞以外の全ての要素をカットオフします。
そのために、まずは画像をグレースケールに変換します。
import cv2
import numpy as np
import matplotlib.pyplot as plt
#pythonでのopencvでは色がBGRで読み込まれることに注意します。
img_gray = cv2.cvtColor(cv2.imread(src),cv2.COLOR_BGR2GRAY)
cv2.imwrite('img_gray.png',img_gray)
cv2.waitKey()
cv2.destroyAllWindows上記の処理で取れたグレースケール画像が以下です。

ヒストグラム解析(cell-based)
次にこの画像をヒストグラム解析します。
img_histogram, img_bins = np.histogram(np.array(cv2.imread(src)).flatten(), bins=np.arange(256+1))
plt.plot(img_histogram)
plt.show()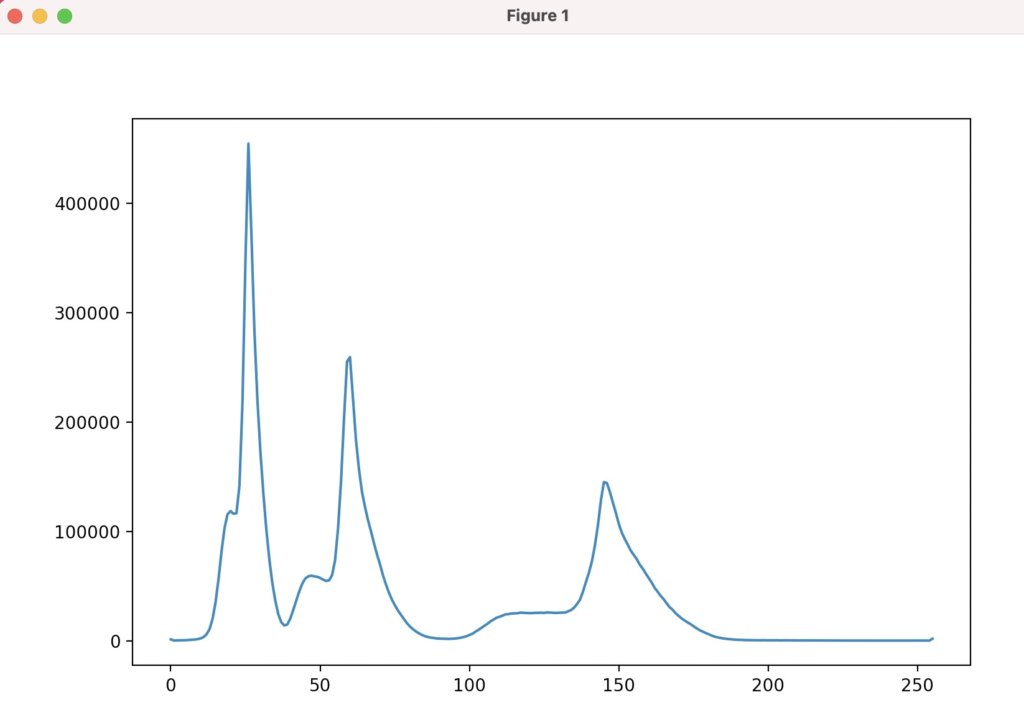
ちょうど二値化の閾値(x軸上)が一番高くなるピークに細胞のプロットが集まっているため、閾値を130から175とします。
upper_th = 175
lower_th = 130
ret, img_th = cv2.threshold(img_gray, lower_th,upper_th, cv2.THRESH_BINARY)
cv2.imwrite('img_binary_cells.png',img_th)
cv2.waitKey()
cv2.destroyAllWindows上記の処理を行うと、以下のように蛍光染色された細胞のみを抽出することができました。

次にゲル中に入っている細胞のみをカウントするために、ゲルのみに着目して二値化を行います。
ゲルの二値化
上記のヒストグラムで挙げたように、細胞のピークが一番右に来ていた時は細胞抽出に都合が良かったですが、ゲルのピークはグレースケール画像中では真ん中に出ています。
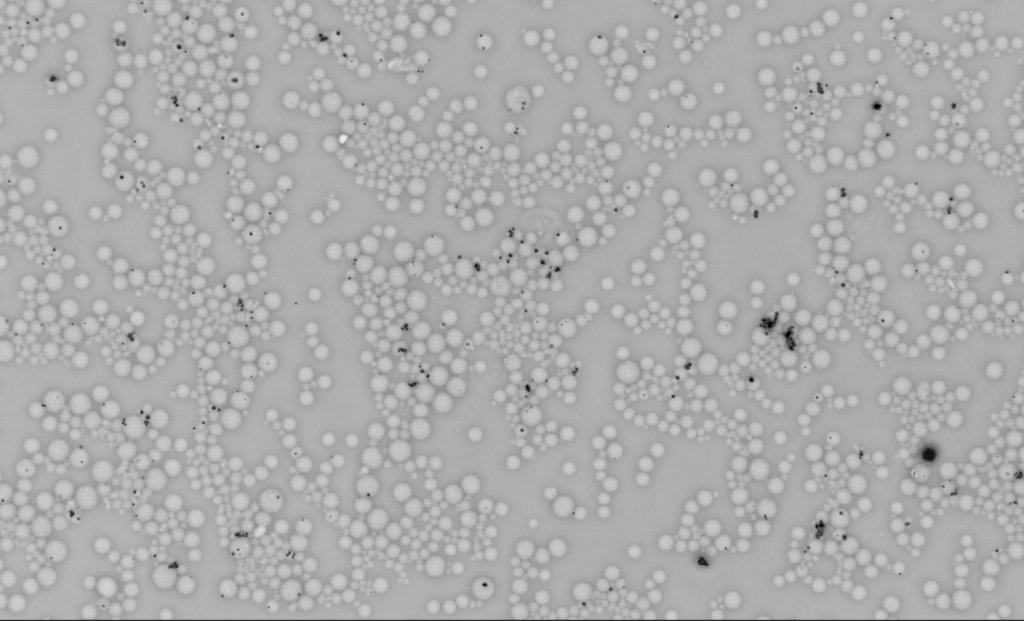
この問題は、グレースケール画像の色を反転させることで解決することができます。
img_gray_reverse = cv2.bitwise_not(img_gray)上記の処理でグレースケールを反転させた画像を取得することができました。
ヒストグラム解析(Gel-based)
続いてこの反転グレースケール画像のヒストグラムを解析します。
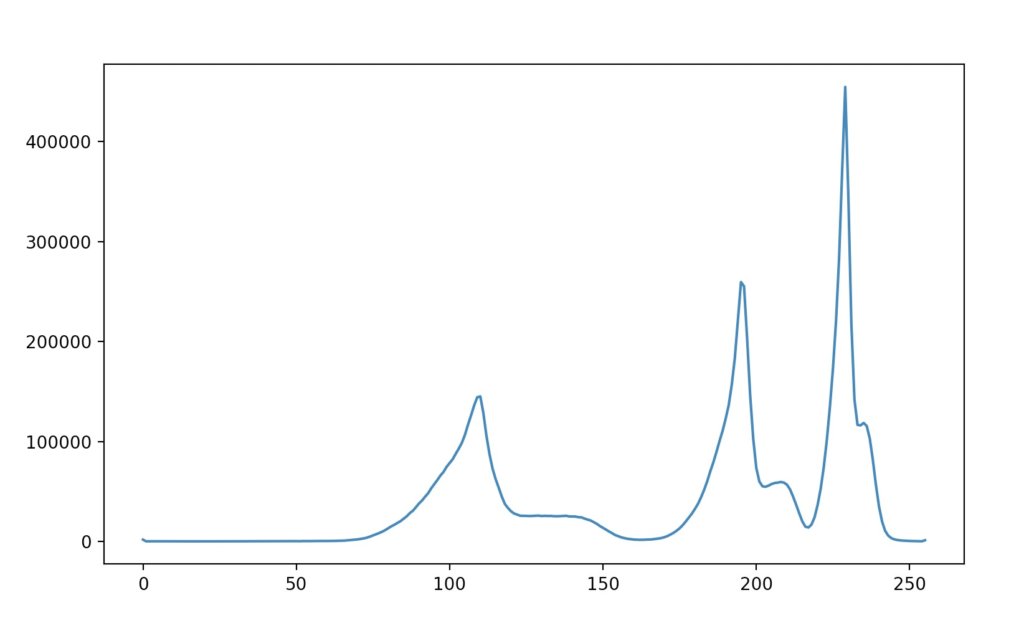
無事反転させることができているようです。
このヒストグラムから、閾値を180から189と定めてみます。
upper_th = 189
lower_th = 180
ret, img_th = cv2.threshold(img_gray_reverse, lower_th,upper_th, cv2.THRESH_BINARY)
cv2.imwrite('img_binary_droplets.png',img_th)
cv2.waitKey()
cv2.destroyAllWindows上記の処理を行うと、ゲルのみに着目した二値化画像を得ることができました。

ゲル内の細胞のみを検出する方法
全ての細胞とゲルを二値化した画像をそれぞれ1つづつ得ることができたため、ゲルを以下の記事のように解析し、その後セルが輪郭中の座標に位置するかを条件分岐で調べていけば、カットオフすべきピクセルの座標が割り出せます。
これにより、ゲル内にトラップされた細胞のみを抽出することが可能となります。



