今回はopenCVを利用して、顕微鏡画像から細胞数をカウントするプログラムを書いてみようと思います。(ImageJでもできます。)
今回利用する細胞は、EBPRリアクターから採取した微生物です。リン酸をポリリン酸の形で蓄積するため、DAPI染色で黄色の蛍光を発します。
目次
サンプル調整プロトコル
以下にサンプルをリアクターから採取する際に行った調整方法をまとめます。
(1)超音波処理
今回の目的は細胞数のカウントなので、細胞を壊さない程度で出来る限り分散させます。いつもの凝集防止用の超音波より2倍くらい長くしました。
また、細胞が急なpH変化や浸透圧によってびっくりしないように、水ではなくPBS-EDTA溶液中で破砕しました。
(2)フィルター
一回めで小さいポアサイズを使用するとバイオマスが引っかかって詰まるので、20μmで大まかなバイオマスを取り除いてから11μmのフィルターを使用しました。
(3)濃度調整
フィルター後の細胞濃度は5.0*10^8/mlくらいあるので、1000倍に薄めました。
(4)染色
染色にはDAPI(DAPI:DW:SlowFade=1:1:2)を使用しました。
openCVによる細胞数のカウントプログラム
今回はPythonでcvモジュールを用いて画像処理を行います。
(1)輪郭検出用関数
関数名をfindContoursとします。
def findContours(src,n1,n2):
img_gray = cv2.cvtColor(src, cv2.COLOR_BGR2GRAY)
ksize = (n1, n2)
img_gray = cv2.GaussianBlur(img_gray, ksize=ksize, sigmaX=0, sigmaY=0)
ret, c1img = cv2.threshold(img_gray, 0, 255,cv2.THRESH_BINARY | cv2.THRESH_OTSU)
cv2.imshow('Binary', c1img)
contours, tmp = cv2.findContours(image=c1img, mode=cv2.RETR_TREE,method=cv2.CHAIN_APPROX_SIMPLE)
result = cv2.drawContours(image=src, contours=contours,contourIdx=-1, color=(0, 0, 255),thickness=2, lineType=cv2.LINE_AA)
cv2.imshow('Result', result)
for i, cont in enumerate(contours):
rect = cv2.minAreaRect(cont)
box = cv2.boxPoints(rect)
box = np.int0(box)
dst = cv2.drawContours(image=src, contours=, contourIdx=0,color= (255, 255, 0), thickness=2,lineType=cv2.LINE_AA)
cv2.ellipse(dst,rect,(0,255,255),2)
print(len(contours))
return dstcv2.COLOR_BGR2GRAYを第二引数として、画像をグレースケールに変換します。
img_gray = cv2.cvtColor(src, cv2.COLOR_BGR2GRAY)細胞は球形なので、中心に重みが置かれるガウシアンフィルタを掛け、二値化を行います。
変数ksizeを第二引数と第三引数で決定します。(この時、n1,n2は正の奇数で指定します。)
ksize = (n1, n2)
img_gray = cv2.GaussianBlur(img_gray, ksize=ksize, sigmaX=0, sigmaY=0)
ret, c1img = cv2.threshold(img_gray, 0, 255,cv2.THRESH_BINARY | cv2.THRESH_OTSU)
cv2.imshow('Binary', c1img)とりあえずオリジナルの画像と二値化した画像を並べて出力しておきます。
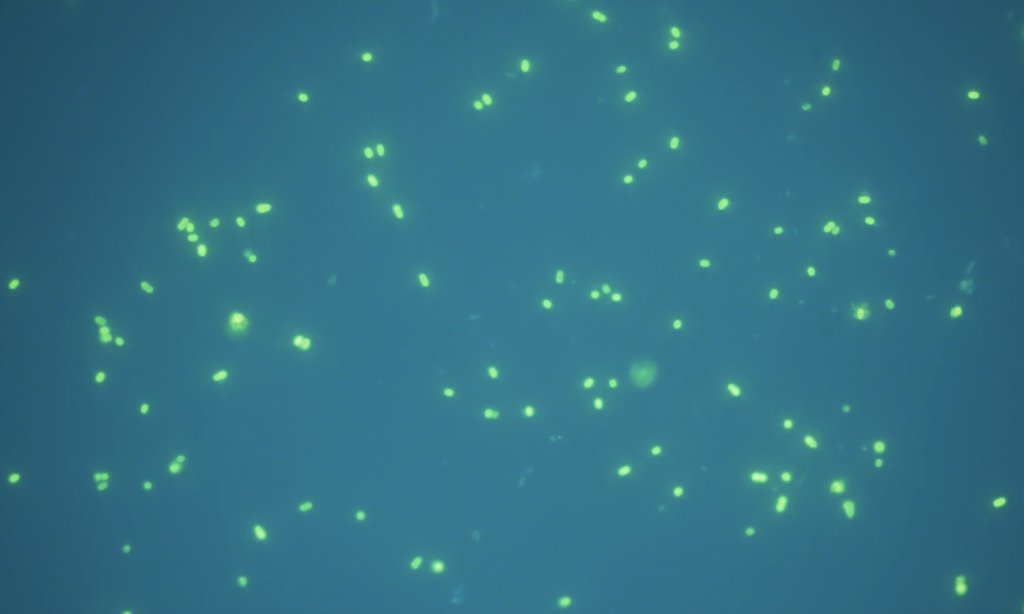

白と黒の2つの値に分けることができたので、上記の画像を元に輪郭を検出していきます。
contours, tmp = cv2.findContours(image=c1img, mode=cv2.RETR_TREE,method=cv2.CHAIN_APPROX_SIMPLE)
result = cv2.drawContours(image=src, contours=contours,contourIdx=-1, color=(0, 0, 255),thickness=2, lineType=cv2.LINE_AA)
cv2.imshow('Result', result)以下が出力された画像です。
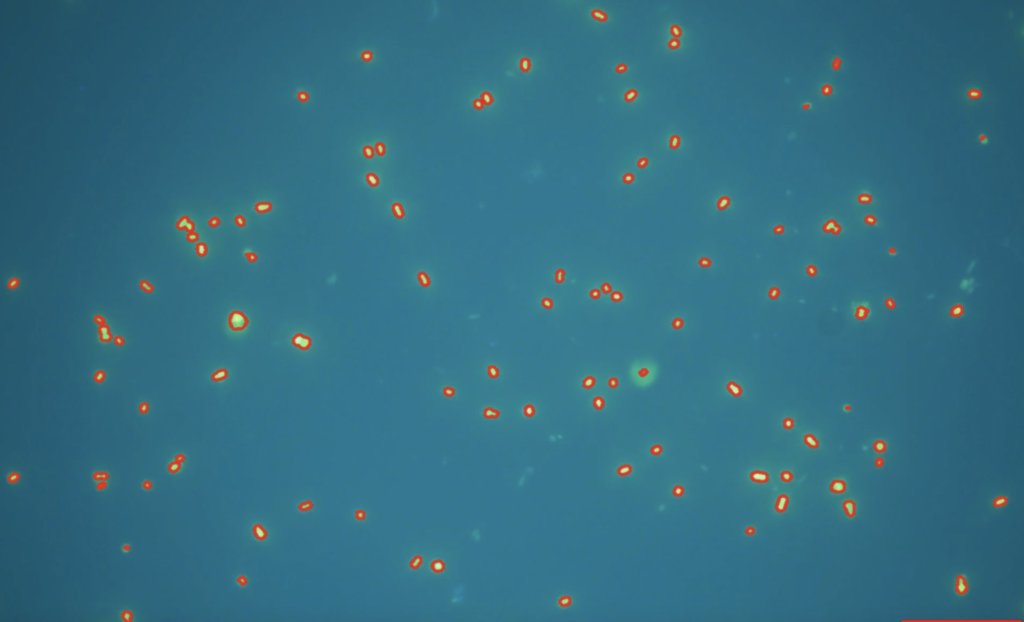
変数contoursの長さが輪郭の数(細胞数)に相当するので、len(contours)を取り出します。
print(str(len(contours))+'cells')(2)画像の下処理を行う関数resizeの定義
以下に関数resizeを定義します。
def resize(src, bsize):
basePixSize = bsize
height = src.shape[0]
width = src.shape[1]
largeSize = max(height, width)
resizeRate = basePixSize / largeSize
dst = cv2.resize(src, (int(width * resizeRate), int(height * resizeRate)),interpolation=cv2.INTER_CUBIC)
return dst(3)処理の実行
main.pyにて以下の処理を実行します。(上記で作成した関数を実行するコードです)
ksizeは25,25としました。
import cv2
from functions import getResize,getFindContours
from source import src
n1 = 25
n2 = 25
if __name__ == '__main__':
dst = resize(cv2.imread(src), 1280)
cv2.imshow("Original", dst)
dst = findContours(dst,n1=n1,n2=n2)
cv2.waitKey(0)
cv2.destroyAllWindows()今回はDAPI染色した単一の細胞がターゲットでしたが、二値化のパラメータを調整することで、特定の蛍光波長のみを抽出することもできます。



