以下の記事で特殊なゲル球にトラップされた細胞の画像を分析しましたが、ここではさらに踏み込んでゲルの外側にいる細胞と内側にいる細胞を分けてみようと思います。
目次
ノイズ除去

画像データを二値化したものですが、円周に凹凸があったり小さなノイズが入っているのがわかります。
これではゲル球を検出しづらいので、ブラーをかけていきます。
(1)中央値ブラー
一番一般的な平滑化は中央値ブラー(MedianBlur)を用いて行うことができます。
import cv2
img = cv2.imread('mg_binary.png')
img_masked = cv2.medianBlur(img,ksize=5)
cv2.imwrite('img_median_blured.png',img_masked)カーネルサイズは奇数で指定します。


輪郭の凹凸がすり減っているのがわかります。
(2)ガウシアンブラー
中央値ブラーがカーネル中の全画素の中央値を使用していたのに対し、ガウシアンブラーでは重み付けの方法を変えたガウシアンフィルタを使います。
import cv2
img = cv2.imread("img_binary.png")
img_masked= cv2.GaussianBlur(img,(5,5),0)
cv2.imwrite('img_gaussian_blured.png',img_masked)

輪郭の検出
上記で作成した図2を使用して、輪郭の検出をしていきます。(今回は中央値フィルターの方が良かったです。)
検出にはfindContoursメソッドを使用します。
import cv2
img = cv2.imread('img_median_blured.png')ここで、findContoursはCV_8UC1形式の画像しか読み込まないため、一旦グレースケールに戻してから閾値を適当に指定してthに代入します。
ret, th = cv2.threshold(cv2.cvtColor(img,cv2.COLOR_BGR2GRAY), 110, 255, cv2.THRESH_BINARY)ここで、findContoursメソッドを使用してcontoursにリスト型の輪郭情報を渡します。
ここで、ゲル球の面積に応じてカットオフできるように新たな変数contours_by_areaを作成し、面積の上限および下限を指定します。
contours, hierarchy = cv2.findContours(th, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_TC89_L1)
contours_by_area = list(filter(lambda x: cv2.contourArea(x) >= 180 and cv2.contourArea(x) <= 1000, contours))contours_by_areaに輪郭情報が格納されているので、ループを使用して取り出していきます。
for i, c in enumerate(contours):
area = cv2.contourArea(c)
print(f"{i}, area{i}: {area}")最後に輪郭を線で囲って元画像と重ね合わせます。
さらに、contours_by_area中の要素数を数えることで、指定面積範囲内のゲル球の個数を算出します。
result = cv2.drawContours(img, contours_by_area, -2, (0,0,254), 1)
cv2.imwrite('img_contours.png',result)
print(len(contours_by_area))上記の処理で取得した画像が以下です。
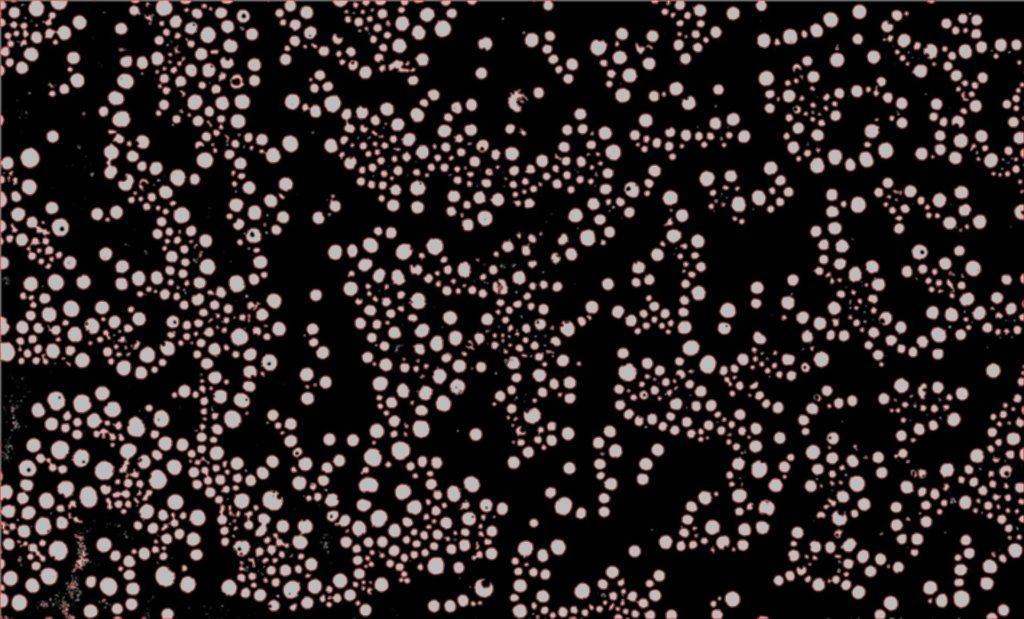
細胞の輪郭検出
上記のゲルの輪郭抽出と同じ要領で、以下の細胞のみを検出した二値化画像の中で輪郭検出を行なっていきます。

ここで問題になるのが、コロニーサイズが大きすぎるものを取り除かなければならない点です。
この問題は面積指定で解決できます。

無事に検出できているのがわかりますね。
今回は面積を0から250に指定し、輪郭線の色を緑色にしました。
これらの画像を重ね合わせることで、特殊ゲル中にトラップされた細胞のみの検出ができそうです。



