大学2年生で習ったミカエリスメンテン式を改めて復習します。
目次
ミカエリスメンテン式
酵素反応はE(enzyme),S(substrate),P(product)を用いて以下のように表されます。
$$E+S\overset{k_{1}}{\underset{k_{-1}}{\rightleftharpoons}}ES\xrightarrow{k_2} P+E$$
ここで、Pの生成速度は
$$v_p=\frac{d[P]}{dt}=k_2[ES]\tag{1}$$
酵素基質複合体の生成速度は
$$\begin{align}v_{ES}&=\frac{d[ES]}{dt}\\&=k_1[E][S]-k_{-1}[ES]-k_2[ES]\quad (2)\end{align}$$
この時、以下の条件を満たす場合、酵素基質複合体は定常状態であると仮定できます。
条件1:酵素基質複合体の生成反応は平衡状態にある。
条件2:存在する全酵素は複合体を形成しており、基質濃度は酵素濃度よりも十分高い。
この時
$$\frac{d[ES]}{dt}=0\tag{3}$$
が成り立ちます。
(2)(3)式より
$$k_1[E][S]-k_{-1}[ES]-k_2[ES]=0\tag{4}$$
酵素の全濃度について
$$[E]_0 = [E]+[ES]\tag{5}$$
より、
$$\begin{align}[ES]&=\frac{[E]_0 [S]}{\frac{k_{-1}-k_2}{k_1}+[S]}\\&=\frac{[E]_0 [S]}{K_m+[S]}\tag{6}\end{align}$$
反応初期の微小時間中では基質と酵素が結合していないため、反応初速度を以下のようにすれば、
$$v=k_2[ES]$$
(6)式より
$$\begin{align}v&=\frac{k_2 [E]_0 [S]}{K_m + [S]}\\&=\frac{V_{max}[S]}{K_m+[S]}\tag{7}\end{align}$$
酵素の反応速度を得る。
Km値の意味
Km値はミカエリス定数と呼ばれます。
例えばKmが小さい場合、酵素の反応速度は大きくなります。つまり、基質との親和性が高いということができます。
Vmax
反応の最大速度であるVmaxは(7)式から
$$\lim_{[S] \to \infty}\frac{V_{max}[S]}{K_m+[S]}=V_{max}$$
となる。
Km=[S]のとき
(7)式について、Km=[S]となるとき、
$$v=\frac{V_{max}}{2}$$
となります。
Pythonで描画
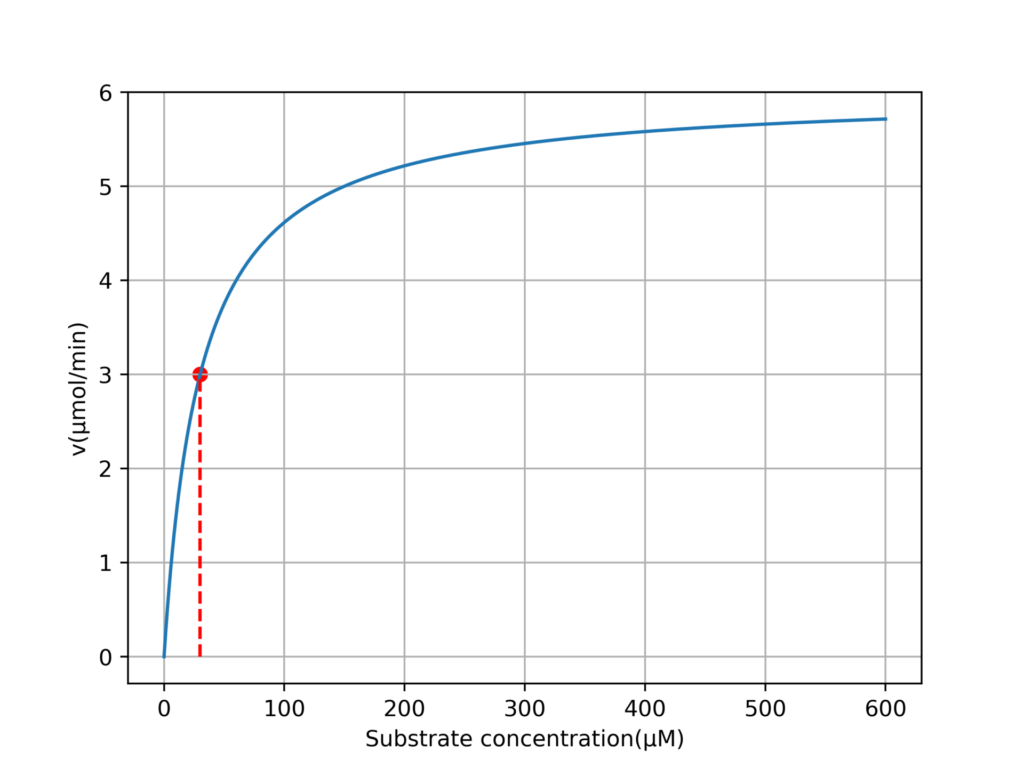
上記の式を踏まえて、具体的なグラフを作成してみます。(実際は計測データからKmとVmaxを求めることになりますが、これらのパラメータが決定していないとグラフ作成ができないため、今回は任意の値を指定します。)
Vmax=6(μmol/min),Km=30 (μM)の場合について考えます。
import numpy as np
import matplotlib.pyplot as plt
fig = plt.figure()
Km = 30
Vmax = 6
S = np.linspace(0,600,1000)
v = (Vmax*S/(Km+S))
plt.plot(S,v)
plt.grid()
plt.ylabel('v(μmol/min)')
plt.xlabel('Substrate concentration(μM)')
plt.vlines(Km,0, Vmax/2,color='red',linestyle = "dashed")
plt.scatter(Km,Vmax/2,color='red')
fig.savefig('result_mica.png',dpi=500)
出力結果