最近OpenCVでの画像解析にハマり、画像の調整からhough変換まで一通りの操作をこなしてきました。
もちろん、GUIベースのImageJでも同じようなことができるのですが、OpenCVの強みはなんといっても画像認識にCNN(畳み込みニューラルネットワーク)を使うことができる点ではないでしょうか。
また、画像の連番入力等の大量のデータを自動で扱える点や、C ++を使用した高速行列計算ができることを考えると、圧倒的にOpenCVの方が優れていると言えるのではないでしょうか。
ただし、CUIベースであることと、実行環境にやや難ありではあります。
例えばCUIベースであることは、作ったプログラムを他の人に使ってもらうときにパラメータを直接変数に代入する形で行わなければいけないことを意味します。
さらに、OpenCVはpythonといったプログラミング言語のモジュールとして動作するので、実行環境にpython等の言語がインストールされている必要があります。
(この辺はDocker等を使用してwebアプリ化できないか検討しています。)
というわけで、今回はアプリ開発といいつつ、.pyファイルを作成していく似非アプリ開発を行なっていこうと思います。
目次
アプリ開発概要
初めに、フローチャートを作成しました。
目的は、画像中の特殊ゲル中にトラップされた細胞を検出することです。
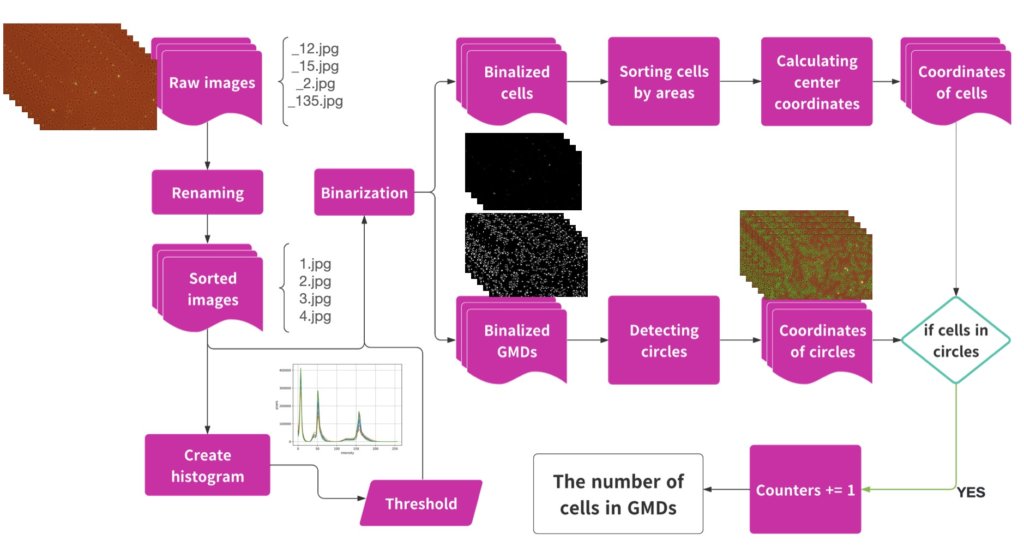
矢印の開始点にあるように、初めはDatasetに解析対象の画像を大量に入れていくのですが、画像の名前がそれぞれ違うという問題が発生します。(画像を順番に読み込みたいときに、名前に一貫性がないと大変です。)
もちろん、画像には作成日時が添付されているのでそれを呼び出すこともできますが、コードを書くのがめんどくさくなるのと、後から見たときにわかりやすくなるように、リネームをします。
これに対抗するために、まずはファイル内の画像を自動的に連番でリネームしてくれるプログラムを作成します。
rename.pyの作成
まずはdataset中にあるdataset_rawの中に解析対象の画像を全て入れておきます。
その後に、dataset_rawの一つ上の階にrename.pyを作成します。
rename.pyの中身
以下が名前を自動的に変更してくれるプログラムです。
返り値はいらないので、returnは空にしておきます。
import os
import glob
def rename():
current_directory = os.getcwd()
dir_rawdata = current_directory + '/data_raw'
print(sum(os.path.isfile(os.path.join(dir_rawdata, name)) for name in os.listdir(dir_rawdata)))
files = glob.glob(str(dir_rawdata)+"/*.jpg")
i = 1
for name in files:
new_name = "{}.jpg".format(str(i))
os.rename(name, new_name)
print(name + "=>" + new_name)
i += 1
if __name__ == '__main__':
rename()今回このプログラムではosとglobと呼ばれるモジュールを使いました。
osはpython実行環境のパスを拾ってきてくれます。
また、globを使うことで特定のパターン(今回は*.jpg)に当てはまるファイルを網羅的に拾ってきてくれます。アスタリスクはpythonにおいて、何でもいいよという意味を持っています。(ワイルドカード)
main.pyの作成
このブログは自分へのリマインド用なので、とりあえず一気にメインファイルのプログラムを貼っておきます。
import cv2
import numpy as np
import matplotlib.pyplot as plt
from tqdm import tqdm
import time
import os
import glob
from PIL import Image
fig = plt.figure()
#denoising parameters
denoising_threshold = 30
#img
cells_binarise_thres_lower = 150
cells_binarise_thres_upper = 188
#img_reverse
gmds_binarise_thres_lower = 187
gmds_binarise_thres_upper = 208
#detecting circles parameters
mindist = 12
p1 = 40
p2 = 10
r_max = 20
r_min = 3
radius_flexibility = 2
#get files from dataset
current_directory = os.getcwd()
dir_rawdata = current_directory + '/dataset_resized'
len_dataset = sum(os.path.isfile(os.path.join(dir_rawdata, name)) for name in os.listdir(dir_rawdata))
filenames = ['{}.jpg'.format(i) for i in range(1,len_dataset+1)]
print('Loading files......')
for i in filenames:
print('Filename:' + i)
for j in tqdm(range(len(filenames))):
time.sleep(0.011)
print('Loading comlete....')
print('######################################################################################')
########################################################
files = os.listdir('dataset/')
for file in files:
if '.jpg' in file:
img = Image.open(os.path.join('dataset/',file))
im_crop = img.crop((0, 0,1900,1150))
im_crop.save('dataset_resized/{}'.format(file), quality=100)
def denoise(img_name,k,denoise_th):
im = img_name
gray = cv2.cvtColor(im,cv2.COLOR_BGR2GRAY)
bimg=cv2.adaptiveThreshold(gray, 255,cv2.ADAPTIVE_THRESH_GAUSSIAN_C, cv2.THRESH_BINARY_INV,11,2 )
contours,hierarchy = cv2.findContours( bimg, cv2.RETR_EXTERNAL | cv2.RETR_TREE , cv2.CHAIN_APPROX_NONE)
new_contours=[]
FILL_COLOR=(0,0,0)
AREA_MAX= denoise_th
i=0
for c in contours:
s=abs(cv2.contourArea(c))
if s <= AREA_MAX:
new_contours.append(c)
cv2.drawContours( im, new_contours, -1,FILL_COLOR,-1)
cv2.imwrite("GMDs_denoised/denoised_GMDs{}.jpg".format(k+1),im)
def get_locations_of_cells(img):
img_gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, thresh = cv2.threshold(img_gray, 120, 255, cv2.THRESH_BINARY)
contours, hierarchy = cv2.findContours(thresh, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_TC89_L1)
contours_by_area = list(filter(lambda x: cv2.contourArea(x) >=0 and cv2.contourArea(x) <= 260, contours))
cells_locations = []
for i,c in enumerate(contours_by_area):
M = cv2.moments(c)
cont = contours_by_area[i]
if len(cont) > 2 and M["m00"] != 0 :
x = M["m10"] / M["m00"]
y = M["m01"] / M["m00"]
cells_locations.append(np.array([x,y]))
elif len(cont) == 2:
x = (int(cont[0][0][0]) + int(cont[1][0][0]))/2
y = (int(cont[0][0][1]) + int(cont[1][0][1]))/2
cells_locations.append(np.array([x,y]))
elif len(cont) == 1:
x = cont[0][0][0]
y = cont[0][0][1]
cells_locations.append(np.array([x,y]))
return cells_locations
def validation(x,y,radius,p,q):
if ((x-p)**2 + (y-q)**2)< radius**2:
return True
def get_matchings(cells_locations):
matching = 0
for i in range(len(cells_locations)):
for j in range(len(circles[0])):
cell_loc_x = cells_locations[i][0]
cell_loc_y = cells_locations[i][1]
p = circles[0][j][0]
q = circles[0][j][1]
radius = circles[0][j][2]
if validation(x=cell_loc_x,y=cell_loc_y,radius=radius,p=p,q=q):
matching += 1
return matching
print('Binarising images...')
for i in tqdm(range(len(filenames))):
#Cells
img = cv2.imread('dataset_resized/{}.jpg'.format(i+1))
img_gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, img_th = cv2.threshold(img_gray,cells_binarise_thres_lower,cells_binarise_thres_upper, cv2.THRESH_BINARY)
cv2.imwrite('cells_binaries/cells_binary{}.jpg'.format(int(i+1)),img_th)
##############################################################################################################################
#GMDs
img = cv2.imread('dataset_resized/'+ str(filenames[i]))
img_r = cv2.bitwise_not(img)
img_gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
img_gray_reverse = cv2.bitwise_not(img_gray)
ret, img_th = cv2.threshold(img_gray_reverse,gmds_binarise_thres_lower,gmds_binarise_thres_upper, cv2.THRESH_BINARY)
cv2.imwrite('GMDs_binaries/GMDs_binary_{}.jpg'.format(int(i+1)),img_th)
##############################################################################################################################
img_hist, img_bins = np.histogram(np.array(img).flatten(), bins=np.arange(256+1))
plt.plot(img_hist)
#img_hist, img_bins = np.histogram(np.array(img_r).flatten(), bins=np.arange(256+1))
#plt.plot(img_hist)
##############################################################################################################################
#print('Binarising progress: ' +str(round((i+1)/len(filenames),2)*100) + '%')
print("Binarising Done")
print('######################################################################################')
#denoise GMDs_binary_i
print('Denoising images...')
for i in tqdm(range(len(filenames))):
img = cv2.imread('GMDs_binaries/GMDs_binary_{}.jpg'.format(int(i+1)))
img_blured = cv2.blur(img,(5,5))
denoise(img_blured,i,denoise_th=denoising_threshold)
#print('Denoising progress: ' +str(round((i+1)/len(filenames),2)*100) + '%')
print("Denoising Done" )
print('######################################################################################')
#getting the locations of cells
cells_locations_list = [get_locations_of_cells(cv2.imread('cells_binaries/cells_binary{}.jpg'.format(i+1))) for i in range(len(filenames))]
#detecting circles
print('Detecting circles..')
circles_num = 0
cells_in_gmds_list = []
for i in tqdm(range(len(filenames))):
img = cv2.imread('GMDs_denoised/denoised_GMDs{}.jpg'.format(i+1))
img_raw = cv2.imread('dataset_resized/{}.jpg'.format(i+1))
img = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY)
circles = cv2.HoughCircles(img,cv2.HOUGH_GRADIENT,1,mindist,param1=p1,param2=p2,minRadius=r_min,maxRadius=r_max)
if circles is not None:
circles = np.uint16(np.around(circles))
circles_num += len(circles[0])
cells_in_gmds_list.append(get_matchings(cells_locations=cells_locations_list[i]))
for k in circles[0,:]:
cv2.circle(img_raw,(k[0],k[1]),k[2]+radius_flexibility,(255,0,0),2)
cv2.circle(img_raw,(k[0],k[1]),2,(0,0,255),1)
#print('Detecting circles progress: ' +str(round((i+1)/len(filenames),2)*100) + '%')
cv2.imwrite('GMDs_circles/GMDs_circles{}.jpg'.format(i+1),img_raw)
print('Detecting circles Done')
print('######################################################################################')
k = 1
print('The number of cells in GMDs')
for i in cells_in_gmds_list:
print('{}.jpg: {}'.format(k,i))
k +=1
print('Average GMDs number:' + str(circles_num/len(filenames)))
plt.xlabel("intensity")
plt.ylabel("pixels")
plt.grid()
fig.savefig("img_histogram.png",dpi = 500)
plt.show()初めに、今回使うモジュールを総動員しときます。
また、画像のヒストグラム解析用にインスタンスを生成しておきます。
import cv2
import numpy as np
import matplotlib.pyplot as plt
from tqdm import tqdm
import time
import os
import glob
from PIL import Image
fig = plt.figure()denoise関数
一定の面積以上または以下の領域のオブジェクトを削除(正確には背景色で塗りつぶす)する関数を定義します。
def denoise(img_name,k,denoise_th):
im = img_name
gray = cv2.cvtColor(im,cv2.COLOR_BGR2GRAY)
bimg=cv2.adaptiveThreshold(gray, 255,cv2.ADAPTIVE_THRESH_GAUSSIAN_C, cv2.THRESH_BINARY_INV,11,2 )
contours,hierarchy = cv2.findContours( bimg, cv2.RETR_EXTERNAL | cv2.RETR_TREE , cv2.CHAIN_APPROX_NONE)
new_contours=[]
FILL_COLOR=(0,0,0)
AREA_MAX= denoise_th
i=0
for c in contours:
s=abs(cv2.contourArea(c))
if s <= AREA_MAX:
new_contours.append(c)
cv2.drawContours( im, new_contours, -1,FILL_COLOR,-1)
cv2.imwrite("GMDs_denoised/denoised_GMDs{}.jpg".format(k+1),im)FILL_COLORはBRGで色情報を入力します。
解析対象もろとも消してしまわないように、denoise_thの値は慎重に決めなければいけません。
細胞の重心計算
以下の記事にまとめていますが、内外判定を行いたい場合、オブジェクトの位置をベクトルで表すことが重要になってきます。
そこで、対象のオブジェクト(細胞)の輪郭から直交座標上の重心座標を算出します。
def get_locations_of_cells(img):
img_gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, thresh = cv2.threshold(img_gray, 120, 255, cv2.THRESH_BINARY)
contours, hierarchy = cv2.findContours(thresh, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_TC89_L1)
contours_by_area = list(filter(lambda x: cv2.contourArea(x) >=0 and cv2.contourArea(x) <= 260, contours))
cells_locations = []
for i,c in enumerate(contours_by_area):
M = cv2.moments(c)
cont = contours_by_area[i]
if len(cont) > 2 and M["m00"] != 0 :
x = M["m10"] / M["m00"]
y = M["m01"] / M["m00"]
cells_locations.append(np.array([x,y]))
elif len(cont) == 2:
x = (int(cont[0][0][0]) + int(cont[1][0][0]))/2
y = (int(cont[0][0][1]) + int(cont[1][0][1]))/2
cells_locations.append(np.array([x,y]))
elif len(cont) == 1:
x = cont[0][0][0]
y = cont[0][0][1]
cells_locations.append(np.array([x,y]))
return cells_locations返り値をcells_locationとして後から使用します。
内外判定
上記の細胞の座標と、細胞を囲んでいる円の両方の座標を比べて内外判定を行います。
def validation(x,y,radius,p,q):
if ((x-p)**2 + (y-q)**2)< radius**2:
return True
def get_matchings(cells_locations):
matching = 0
for i in range(len(cells_locations)):
for j in range(len(circles[0])):
cell_loc_x = cells_locations[i][0]
cell_loc_y = cells_locations[i][1]
p = circles[0][j][0]
q = circles[0][j][1]
radius = circles[0][j][2]
if validation(x=cell_loc_x,y=cell_loc_y,radius=radius,p=p,q=q):
matching += 1
return matchingmatching で帰ってきた数が、ゲル中にトラップされている細胞の個数になります。
処理の可視化
このアプリでは処理が5個以上ありますが、それらの結果を実行中に確認することは難しいです。(plt.imshow()は同時に一度しか使えない。ため)
そのため、処理の途中で出てきた画像を一つにまとめたものが処理ごとにあると便利です。
今回は13個のサンプルデータを使用していますが、これらを全て処理ごとにまとめて見たいと思います。
import time
import cv2
import numpy as np
import matplotlib.pyplot as plt
from tqdm import tqdm
import math
from PIL import Image
#get files from dataset
import os
import glob
current_directory = os.getcwd()
dir_rawdata = current_directory + '/dataset_resized'
len_dataset = sum(os.path.isfile(os.path.join(dir_rawdata, name)) for name in os.listdir(dir_rawdata))
filenames = ['{}.jpg'.format(i) for i in range(1,len_dataset+1)]
def get_resultimg(img_name,save_to):
imgnum = len(filenames)
fig =plt.figure(figsize=(20,20))
for i in tqdm(range(len(filenames))):
img_name_i = img_name + str(i+1) + '.jpg'
time.sleep(0.0012)
img_i = Image.open(img_name_i)
plt.subplot(math.ceil(imgnum / 3),3,i+1)
plt.imshow(img_i)
print('processing image..../')
fig.savefig(save_to,dpi=500)
get_resultimg("dataset_resized/","results/dataset_resized_all.jpg")
get_resultimg("GMDs_binaries/GMDs_binary_","results/GMDs_binaries_all.jpg")
get_resultimg('GMDs_circles/GMDs_circles','results/GMDs_circles__all.jpg')
print('processing')
get_resultimg('GMDs_denoised/denoised_GMDs','results/denoised_GMDs__all.jpg')
get_resultimg('cells_binaries/cells_binary','results/cells_binaries_all.jpg')
print('#################################################################################################################')
print('processing done!')CUIであるため、処理待ちの時間が味気なくなりがちですので、tqdmモジュールでターミナルを賑わせます。



